[caption id="attachment_327045" align="aligncenter" width="300" caption="Ilustrasi: Metagenomik, teknologi generasi ketiga yang bisa mengeksplorasi semua mikroba dalam suatu sampel"][/caption]
Teknologi diagnosis penyakit menular di Indonesia sudah tertinggal hampir 10 tahun dibanding negara-negara maju, termasuk dari negara tetangga, seperti Singapura dan Malaysia. Di Indonesia, untuk mendiagnosis penyakit menular, dokter masih bergantung pada hasil pengamatan mata peneliti laboratorium klinik. Padahal kini dua generasi teknologi sudah berkembang luas di luar negeri, yakni teknologi DNA sequencing berbasis kultur dan non kultur.
Teknologi “usang” yang masih diterapkan di Indonesia tidak lagi menjadi tumpuan utama mendiagnosis penyakit di beberapa negara maju dan berkembang karena dianggap tidak akurat sehingga berpotensi menimbulkan multitafsir bagi dokter. Satu sampel klinis bisa diartikan lain oleh petugas klinis yang berbeda. Akibatnya, dokter yang membaca hasil pengamatan itu bisa mengambil kesimpulan yang salah dan merugikan pasien. Lebih parah lagi, banyak dokter yang bahkan tidak “mendidik” pasien untuk meminta uji klinis sebelum dokter menyimpulkan penyakit pasien. Tidak jarang, dokter sekadar mendengar keluhan pasien lalu mengambil kesimpulan berdasarkan pengalaman dokter tersebut.
Di Indonesia, laboratorium klinis masih terbilang konvensional atau bisa dikatakan sebagai “generasi pertama”. Kuman dari sampel pasien ditumbuhkan (dikultur) lalu dikenali berdasarkan reaksi biokimia dan pengamatan visual. Metode ini masih sangat bergantung pada kejelian mata dan kepakaran ahli mikrobiologi di laboratorium tersebut. Kalau salah menebak, pasien yang menanggung akibatnya. Padahal, tidaklah mudah “menebak kuman dengan benar” karena penampilan kuman seringkali menipu. Satu kuman seringkali memiliki bentuk dan sifat kimia yang susah dibedakan dengan kuman yang lain, contohnya Aerococcus urinae dan Aerococcus sanguinicola yang menyebabkan infeksi saluran kencing. Padahal antibiotik untuk A. urinae tidak mempan untuk A. sanguinicola, begitu pula sebaliknya.
[caption id="" align="aligncenter" width="512" caption="Sequencer: mesin untuk mentransformasi DNA menjadi file berukuran gigabytes"]

Model “jadul” itu di luar negeri perlahan-lahan mulai ditinggalkan. Sudah jamak laboratorium klinik menggunakan teknologi sequencer untuk identifikasi kuman di sampel pasien. Sequencer adalah mesin canggih yang bisa menuliskan deret DNA dari suatu sampel. DNA kuman diproses sequencer untuk diidentifikasi secara akurat. Tampak mata, boleh saja dua kuman memiliki penampakan serupa, tetapi mereka memiliki deret DNA yang berbeda.
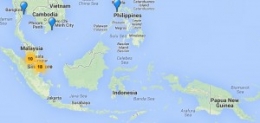
[caption id="attachment_327048" align="aligncenter" width="300" caption="Malaysia punya 10 sequencer, Singapura punya 18, Filipina punya 2, Indonesia mana? (Sumber: http://omicsmaps.com/)"]
Berdasar data dari World Map of High-throughput Sequencers, Indonesia belum memiliki satu pun mesin itu. Indonesia bahkan tertinggal dari Malaysia yang sudah ada 10 sequencer. Singapura yang penduduknya jauh lebih sedikit dari Jakarta bahkan sudah mempunyai 18 sequencer.
Teknologi “generasi kedua” itu memiliki tahapan hampir sama dengan “generasi” pertama yakni sama-sama membuang materi non-DNA dari kuman yang hendak diteliti, lalu petugas laboratorium klinik menumbuhkan berbagai kuman yang ada di sampel tersedia dan memilih satu jenis kuman yang hendak diidentifikasi. Bedanya, pada “generasi kedua”, DNA dari satu jenis kuman itu diproses oleh sequencer yang menghasilkan file deret DNA dalam ukuran gigabytes. Deret tersebut dianalisis, dan akhirnya kuman teridentifikasi. Sederhananya, perbandingan keakuratan diagnosis metode “generasi kedua”dan “generasi pertama” adalah seperti mengukur kekuatan gempa bumi dengan menggunakan seismograf dan sekadar menebak dengan “perasaan”.
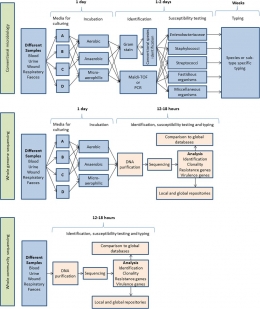
[caption id="attachment_327041" align="aligncenter" width="300" caption="Durasi dari diagnosis menggunakan teknologi generasi pertama (conventional microbiology), generasi kedua (whole genome sequencing), dan generasi ketiga (whole community sequencing)"]
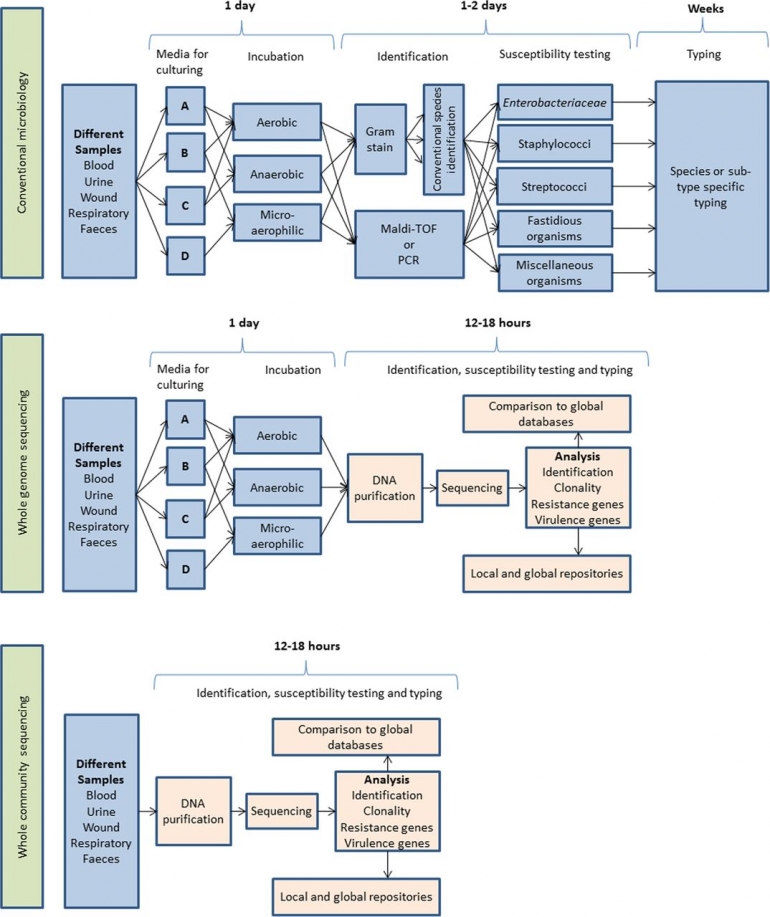
Namun, teknologi “generasi kedua” pun masih memiliki kelemahan. Pertama, jika kuman tidak bisa ditumbuhkan di laboratorium, maka kuman tidak bisa dideteksi oleh sequencer. Kedua, waktu untuk menumbuhkan kuman di laboratorium bisa sangat cepat atau sangat lambat. Bakteri E. coli yang dikenal salah satunya sebagai penyebab diare, bisa ditumbuhkan dalam sehari, tapi menumbuhkan bakteri TBC butuh waktu tiga bulan. Itu pun, tidak selalu berhasil.
[caption id="attachment_327040" align="aligncenter" width="438" caption="Teknologi generasi kedua (atas) vs generasi ketiga (bawah)"]
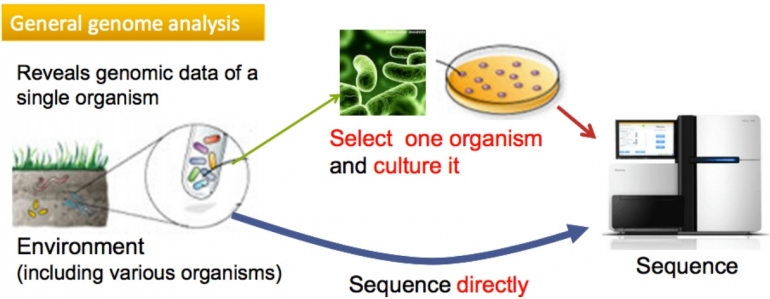
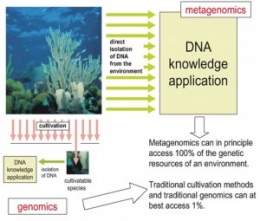
Diagnosis berbasis metagenomik (nonkultur) atau bisa dibilang “generasi ketiga” adalah metode pertama di dunia yang saya dan kolega di Center for Genomic Epidemiology yang berbasis di Denmark perkenalkan melalui Journal of Clinical Microbiology. Metode ini memangkas prosedur penumbuhan kuman di laboratorium, sehingga DNA dari sampel pasien bisa langsung dimasukkan ke sequencer. DNA dari berbagai kuman itu akan ditransformasi ke dalam bentuk file. Berbagai macam kuman yang terdeteksi oleh sequencer akan “dibaca” oleh algoritma analisis yang saya kembangkan. Hasil akhir analisis adalah berupa profil komunitas makhluk hidup dalam sampel itu. Keuntungan bagi pasien, mereka bisa mengetahui penyakit yang mereka derita dalam waktu kurang dari sehari setelah sampel diterima, dengan biaya relatif tidak terlalu jauh berbeda dengan “generasi pertama”. Tentu saja dengan kadar akurasi yang jauh lebih prima. Lebih penting lagi, dengan diagnosis ala “generasi ketiga”, dokter juga bisa tahu bahwa si pasien resisten terhadap antibiotik tertentu.
[caption id="attachment_327046" align="aligncenter" width="300" caption="Hanya 1% dari makhluk hidup dalam sampel yang terdeteksi oleh teknologi generasi pertama dan kedua, tapi 100% makhluk hidup terdeteksi dengan teknologi generasi ketiga"]
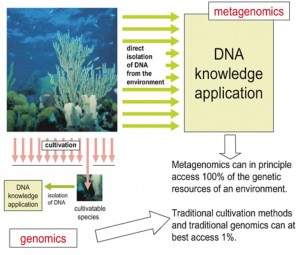
Saat ini penelitian diagnosis berbasis metagenomik yang merupakan bagian dari disertasi doktoral saya, sudah berada pada tahap pembuktian konsep (proof of concept). Tentu saja, sebelum teknologi ini digunakan, diperlukan penelitian ke lebih banyak pasien. Saya sudah menguji algoritma saya, yaitu Chainmapper, ke beberapa studi kasus, dengan hasil sebagai berikut:
- Saya meneliti sampelurin 24 pasien terduga infeksi saluran kencing di sebuah rumah sakit di Kopenhagen pada April dan September 2012. Hasilnya, semua kuman dalam sampel urin itu teridentifikasi. Ada pasien yang kencingnya bebas dari kuman. Ada pasien yang kencingnya mengandung kuman-kuman standar penyebab infeksi saluran kencing. Ada juga pasien yang kencingnya berisi bakteri-bakteri yang dalam buku teks kedokteran tidak pernah dikaitkan dengan infeksi saluran kencing. Namun, beberapa bulan lalu riset menunjukkan bakteri tersebut juga menyebabkan penyakit infeksi saluran kencing. Padahal, kuman tersebut tak bisa ditumbuhkan di laboratorium. Selain itu, ada juga urin yang mengandung bakteri penyebab diare yang biasanya ditemukan di tinja manusia. Boleh jadi, ini karena pasien salah “bersih-bersih” setelah buang air besar atau karena hubungan seks anal.
- Saya berhasil menjabarkan daftar kuman yang menyebar di sebuah rumah sakit, yang dikenal sebagai kuman nosokomial. Infeksi nosokomial menyebar dengan perantara pasien, serangga, atau staf rumah sakit, lalu menyerang siapapun yang tingkat kekebalan tubuhnya rendah. Dengan mengetahui jenis kuman nosokomial, pihak rumah sakit bisa mengantisipasinya. Kita ingat nasihat orang tua, sekembali dari rumah sakit sebaiknya ganti pakaian dan mandi. Ini merupakan salah satu bentuk kearifan lokal. Mereka mungkin belum mengetahui soal kuman nosokomial, tetapi boleh jadi mereka dahulu belajar dari kasus empirik, kenapa orang yang baru pulang dari rumah sakit rentan penyakit atau menyebabkan orang yang berkontak dengan mereka sakit. Dari sisi klinis, hal ini bisa dijelaskan karena kuman nosokomial bisa menempel di baju atau badan lalu ditularkan ke orang lain.
- Penelitian saya, bekerja sama dengan para arkeolog dari Museum Kopenhagen, menguak profil kesehatan penduduk Kopenhagen 300 tahun silam, sekaligus kemungkinan wabah penyakit di masa itu. Penelitian ini berpusat pada sebuah situs jamban purbakala (old latrine) yang diekskavasi mulai musim panas 2011 hingga 2012. Dengan teknologi sequencer, saya bisa menarik kesimpulan bahwa tinja purba tersebut mengandung bakteri-bakteri baik yang umumnya ditemukan pada usus orang yang tidak banyak mengonsumsi karbohidrat, melainkan mengonsumsi makanan sehat kaya serat; mayoritas sayur dan buah. Bakteri-bakteri itu juga bisa menurunkan kandungan kolesterol dalam darah. Berbekal analisis akhir sequencer ditambah observasi lapangan, arkeolog bisa mencapai hipotesis bahwa penduduk Kopenhagen pada masa itu makan makanan sehat. Lebih jauh, mereka bisa menduga pada masa itu Kopenhagen adalah pusat lalu lintas perdagangan buah dan makanan berserat dari berbagai negara.
- Penelitian saya bisa menguak kondisi kesehatan penumpang pesawat terbang secara umum, sekaligus mempelajari kemungkinan kuman yang jadi “penumpang” gelap dari luar negeri. Bekerjasama dengan lima maskapai penerbangan dari kota yang berbeda, saya membandingkan profil kesehatan para penumpang dari lima aviasi tersebut, lebih tepatnya penumpang yang buang air di pesawat-pesawat itu. Saya menemukan bahwa penumpang yang datang dari negara tertentu memiliki komposisi bakteri tinja yang sehat, dan beberapa tidak sehat. Hal itu berkaitan erat dengan pola makan rata-rata penduduk dari kota/negara tersebut. Pada penelitian ini ditemukan juga sejumlah bakteri Ruminococcus gnavus yang diduga membawa wabah penyakit menular dari salah satu negara. Bakteri tersebut sempat dikaitkan dengan penyakit (bacteremia) dari beberapa pasien, bahkan ada pasien tersebut yang meninggal, akan tetapi penelitian ini masih tergolong baru dan belum konklusif, sehingga tingkat keberbahayaannya sampai sekarang masih menjadi misteri.
- Penelitian lain saya adalah membongkar rahasia di balik misteri burung nazar yang tetap sehat kendati memangsa daging mengandung penyakit. Seperti diketahui, burung nazar umumnya memangsa bangkai hewan yang mati karena penyakit menular. Kita juga tahu burung nazar suka makan trenggiling dan biasanya trenggiling menyebarkan penyakit lepra. Apakah burung nazar sebetulnya kena lepra tapi tidak merasakan dampaknya, ataukah hanya perantara wabah? Penelitian ini masih panjang dan berpotensi untuk membantu di bidang kesehatan.
Selain itu, beberapa penelitian nonmedis yang juga memanfaatkan algoritma bioinformatika Chainmapper untuk metagenomik tersebut adalah:
- Memprofil komposisi makhluk hidup di laut kutub, berdasarkan ekspedisi LOMROG II di kutub utara pada tahun 2009 dan ekspedisi Galathea 3 di Tanah Hijau (kutub utara) dan Antartika (kutub selatan) di tahun 2006-2007. Penelitian ini bertujuan salah satunya untuk menguak misteri komposisi kehidupan purba di laut kutub. Dalam palaeontologi dikenal istilah penanggalan endapan danau (lake-sediment dating), yaitu merunut usia benda purbakala berdasarkan kedalaman tempat ditemukannya benda tersebut dan faktor lain dari lingkungan danau tersebut. Tim ekspedisi mengambil sampel laut dari berbagai kedalaman, lalu sekumpulan DNA purbakala tersebut ditransformasi ke dalam bentuk file. Dari situ algoritma saya digunakan untuk memprofil komposisi makhluk laut purba di beberapa kurun.
- Memprofil populasi konifer (pepohonan runjung) di pulau Andøya, Norwegia utara berdasarkan deret DNA purba (ancient DNA). Penelitian ini merupakan bagian dari pembuktian bahwa sekelompok pohon cemara dan pinus Skandinavia ternyata tidak ikut punah dan bahkan tersebar luas di akhir zaman es. Penemuan ini menyanggah teori populer selama ini bahwa pohon-pohon tersebut mati dan punah setelah lapisan es tebal menutupi Skandinavia 9000 tahun silam.
Membeli sequencer merupakan hal yang sangat saya rekomendasikan. Namun, tidak kalah penting, perlu pula mengembangkan cabang ilmu bioinformatika di Indonesia. Saat ini di negara kita, bioinformatika hanya mata kuliah dengan bobot 2 sistem kredit semester pada Jurusan Informatika untuk jenjang sarjana. Padahal, Indonesia merupakan “gudang” penyakit-penyakit tropis yang langka dengan keragaman hayati yang tinggi. Sebagai pembanding, Denmark yang berpenduduk jauh lebih kecil dengan tingkat kerentanan penyakit menular lebih rendah, memiliki banyak pusat penelitian bioinformatika.
Namun, semua gagasan itu tentu bermuara pada dua pertanyaan. Maukah Indonesia berlomba dengan negara-negara maju di bidang bioinformatika? Atau maukah institusi medis Indonesia membantu pasien mendapat hasil diagnosis penyakit lebih akurat lewat teknologi tepat guna semacam metagenomik?
[caption id="" align="aligncenter" width="479" caption="Ilustrasi: Differential diagnosis oleh tim dokter (sumber: Fox)"]

Artikel ini ditulis sebagai tulisan populer dari paper yang penulis publikasikan bersama kolega di Journal of Clinical Microbiology: http://jcm.asm.org/content/52/1/139.short
Penulis: Dhany Saputra Editor: Antony Lee